本文来自微信公众号:西湖大学WestlakeUniversity(ID:westlakeuni),作者:张骊駻实验室,摄影:朱丹阳,编辑:徐珊,校对:张弛,原文标题:《西湖大学张骊駻实验室绘制出世界首张芳香型聚酮全景图》,题图来自:张骊駻实验室
两年前,我们曾经一起敲开过西湖大学理学院张骊駻实验室的大门。这位出生于中国、成长于日本的年轻PI,对大自然如何就地取材,创造出无数能用作药物的天然产物充满了兴趣。因为人类使用的药物近半数都是天然合成物,搞清楚这个创造过程,我们就可以自主、高效、快速地研发出所需的药物。
当时,他瞄准的对象是聚酮类化合物,这是目前已知结构和功能最多样的天然化合物之一,且被广泛应用于药物中。
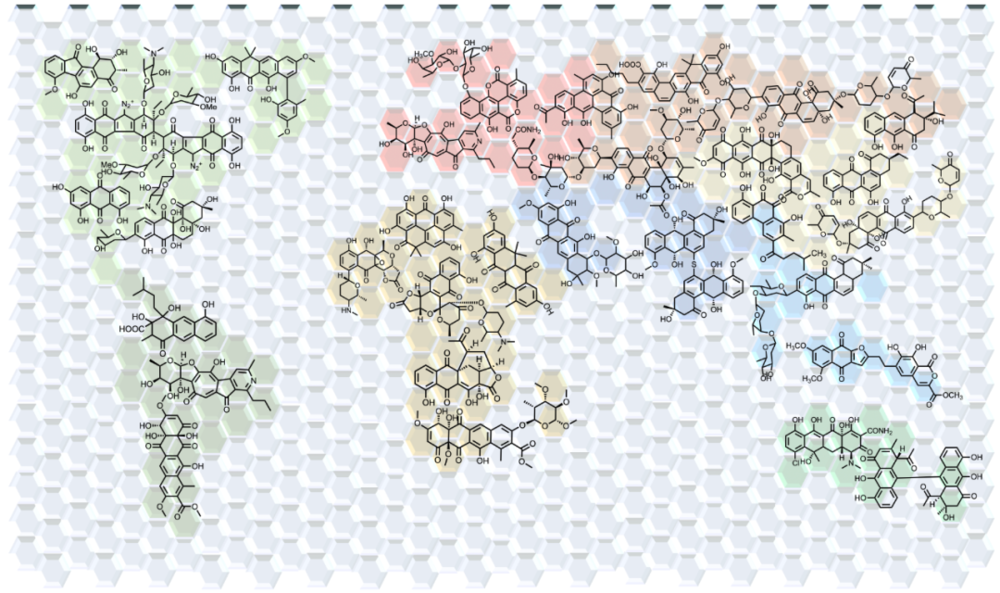
两年之后的今天,张骊駻交出了第一张答卷——他们探究了细菌来源的芳香型聚酮化合物的进化过程及其结构多样性,绘制出世界上首张芳香型聚酮的全景图。正如化学家门捷列夫所贡献的元素周期表,让人类能够有序认识曾经“无序”散乱的化学元素,并据此发现新的物质,这张全景图,也将为我们系统性揭示芳香型聚酮化合物更广博的世界。
近日,该项成果发表于《德国应用化学》(Angewandte Chemie International Edition),论文的通讯作者为西湖大学理学院特聘研究员张骊駻,共同第一作者为西湖大学博士生陈闪冲和助理研究员张驰。

大自然是位奇妙的“造物主”,造就了世界上千千万万形态各异的灵动生命体。从花鸟鱼虫,到飞禽走兽,再到自然界的整体景观……
张骊駻实验室专注研究的,是大自然于微观尺度上的“作品”:天然产物。比如治疗皮炎的红霉素,对付肺炎的氯霉素,应对细菌感染的青霉素……这些耳熟能详的常用药品,都是基于天然产物或天然产物衍生物。
“我们的目标是搞清楚大自然是如何制造出这些天然产物的。“张骊駻解释说,“就像制造一台汽车,我们要搞清楚这条生产线上,工人是谁,部件是哪些,他们是怎么把车身、内饰、轮胎、大灯等组件组装在一起,生产出一台完整的车。”
张骊駻这次挑战的,就是聚酮化合物家族里的一类——细菌来源的芳香型聚酮化合物——它是许多重要药物的核心成分,包括治疗肿瘤的阿霉素和抗菌药四环素,同时因为它的合成“生产线”很特殊、是多个酶协调负责合成产物,“预测”难度非常大。
为了高效揭开这类天然化合物合成的面纱,他们充分发挥了学科交叉的魅力。
“传统的药物发现方法,即从动植物、微生物中,一个一个分离鉴定出新的天然产物,逐个研究学习的方法,只能积累个别案例,无法高效给出答案。但生物信息学,即利用计算机搜索、处理和利用生物学数据的研究方法,给天然产物的发现带来了曙光。”
张骊駻进一步解释道:“现在,我们能够生成、保存和分析海量数据,通过基因组序列数据,锁定天然产物生物合成基因,去预测天然产物分子多样性、丰度、以及其分布——也就是说,更好地了解天然产物的合成机理及它们的‘现状’。”
基于团队的前期研究与资料分析,张骊駻团队认为,芳香型聚酮分子生物合成过程中, CLF(Chain Length Factor;链长因子)这种酶发挥了关键作用。每个聚酮分子都需要这个酶,并且不同类型聚酮分子的CLF酶,会有不同的氨基酸序列。
锁定对象,他们开启了验证之旅。
第一步,研究团队将目光投向167个已经被研究过(即已表征)、已知晓天然产物对应关系的基因簇(即细菌菌株中编码生物合成酶的一列或几类基因)。他们分析发现,这些基因簇中CLF蛋白的氨基酸序列的信息特征,正好对应着不同的化合物的结构特征;换句话说,通过获得CLF的基因信息,我们能够预测最终合成的化合物类别。
同时,同借助CLF这座“桥梁”,研究团队得出了一个能够“拍板”该基因簇是否能产生不同化合物的计算方法及数值线,88%——简单来说,将两个CLF的氨基酸序列作比较,88%以上都一样的话,意味着它们最终合成产物化合物也是一样的;但低于88%,则会产生不同化合物——换言之,我们能够预测最终化合物的“唯一性”。
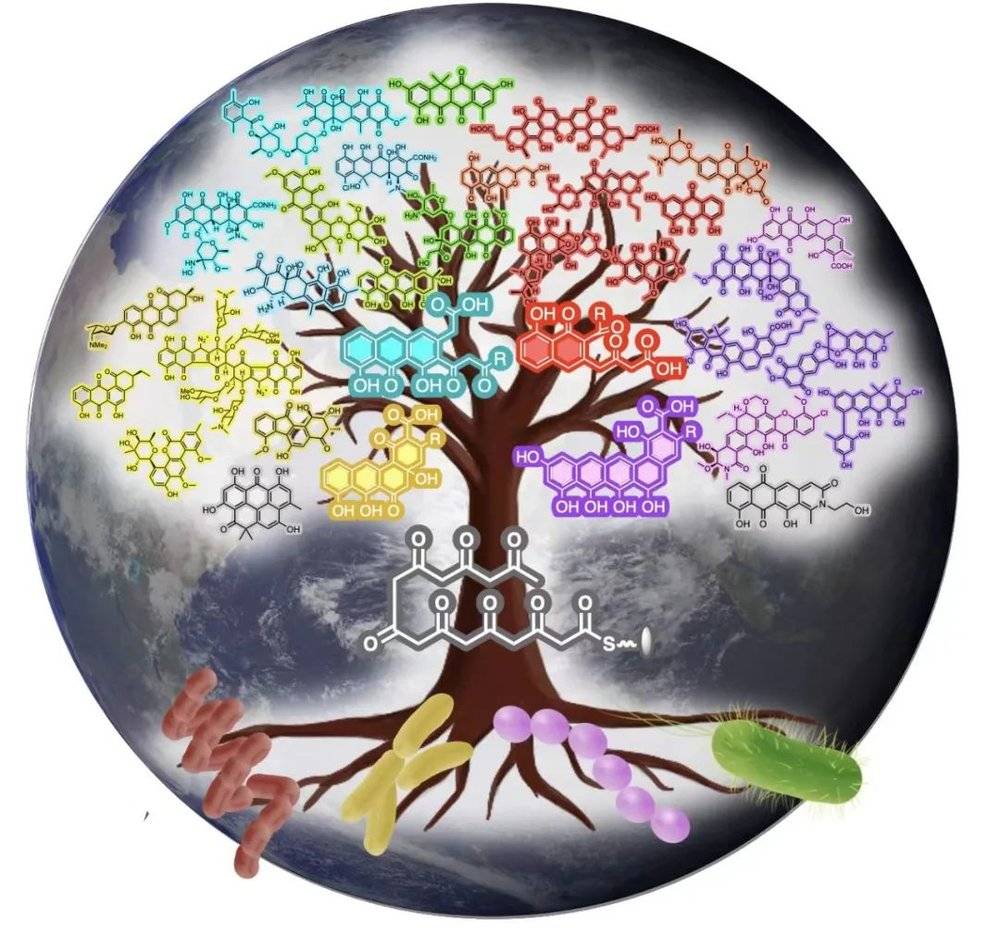
第二步,研究团队从公共基因组数据库中,进一步提取了3254个细菌来源的芳香型聚酮化合物的生物合成酶的数据——这3254个酶,源自现今已被测序的全球所有的微生物(细菌)。接着,他们将第一步的研究办法,应用到了这3254个酶上,构建出了描述“酶氨基酸序列-化合物结构”对应关系的全球“系统发育树”(即图2的细菌来源芳香型聚酮天然产物的全球蓝图)。同时,他们将细菌菌株信息纳入图表(即图表上最外圈的圆环),进而又得到了菌株与化合物之间的对应关系。
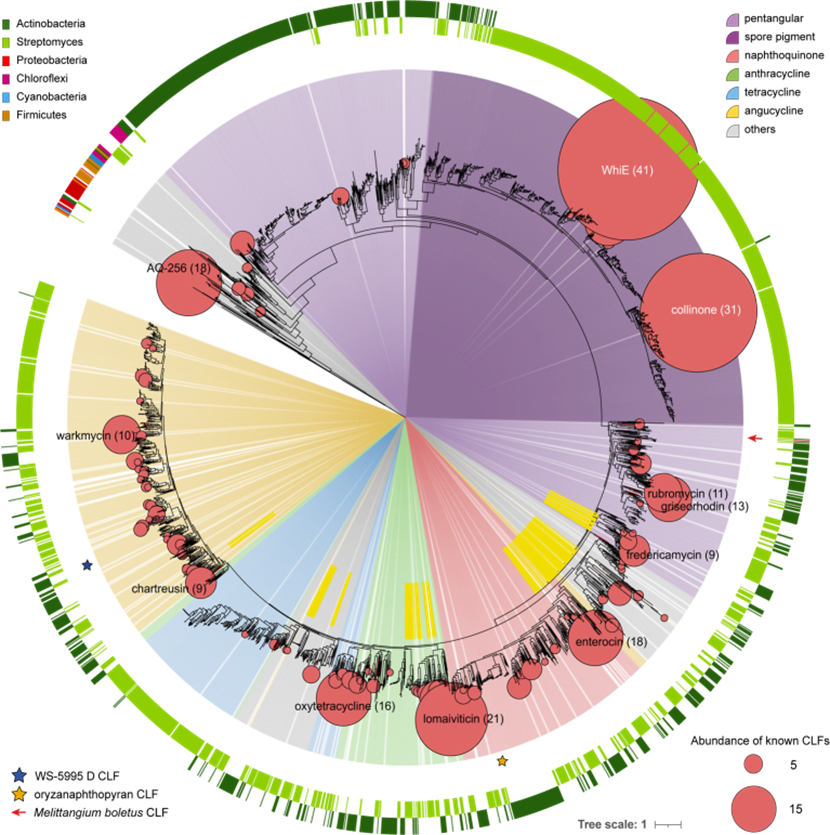
基于这张蓝图式的图表,团队分析了芳香型聚酮的全球丰度、分布和结构多样性。所谓丰度,即每种聚酮化合物在这个“家族”中的丰富程度;分布,则为化合物是在哪些菌里存在;结构多样性,即为这类聚酮化合物,究竟有多少种不同形状。由此,他们预估了自然界的总芳香型聚酮分子数量——也就是细菌来源芳香型聚酮天然产物的“人口数”,大约3000。
一张色彩绚丽的细菌来源芳香型聚酮天然产物全景图,就此诞生。
但激动的团队成员们,依然以科学探究的严谨精神保持了此刻的清醒,再做最后一步验证。他们把这张图表“引入”现实——按图索骥,利用这张芳香型聚酮全球蓝图,看看能否找到新的能够产生新型芳香聚酮化合物的细菌。
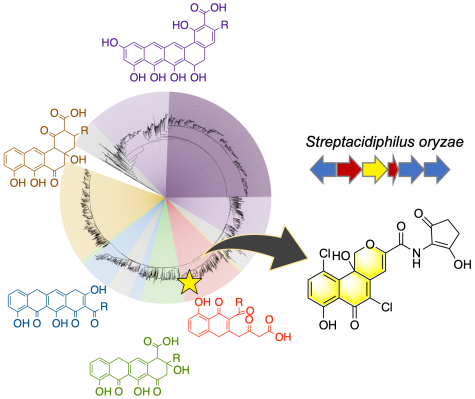
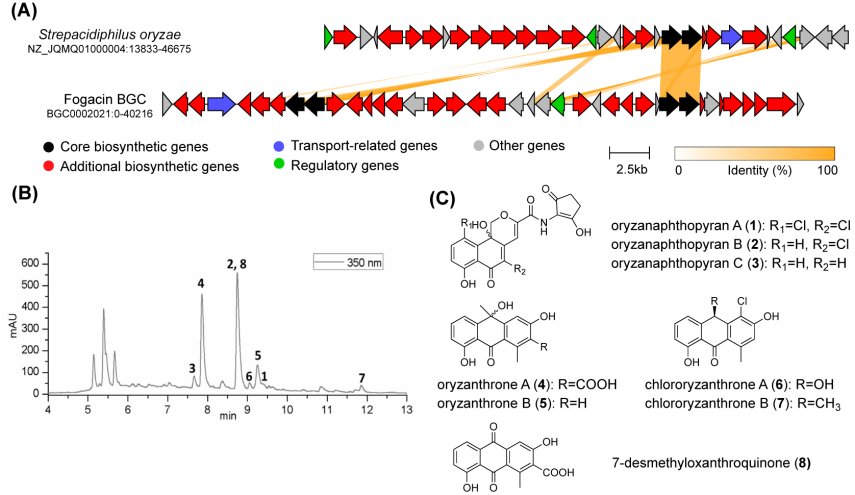
论文一作之一的西湖大学博士生陈闪冲介绍说,研究团队通过这张蓝图和进一步的分析,筛选出了7种菌株;接着,经过进一步分析,锁定了两种能产生新型聚酮化合物的细菌。通过在实验室中进行菌株培养,他们在这两种菌株发酵后形成的最终产物中,均鉴定出了新型的芳香聚酮分子,包括从一株罕见嗜酸放线菌中分离得到了带有新颖骨架的萘吡喃类化合物oryzanaphthopyran。这些发现,最终反过来佐证了本研究所取得的全景图在这类天然产物研究中的实际价值。
回顾两年来的探索历程,张骊駻说:“我们遇到的最大难题是如何把基因信息——我们所已有的数据,大规模地转化为化学信息——即具体的聚酮化合物。基因数据,正如计算机的0和1,可以说是‘数字密码’;如何将它们与聚铜化合物(的结构信息)进行对应,是比较难的。”
总之,这项研究通过基因大数据分析,揭示了细菌来源芳香聚酮的全景图,并参考利用这一宏观地图成功获得了结构新颖的芳香聚酮化合物,极大地提高了对最终合成的化合物预测的精准度。事实上,这是天然产物研究历史上首次达到的分子结构层次分辨率的全球分析——“如果说以前的研究是从远处判断路上的车是什么颜色,我们的研究,相当于可以预测是什么牌子的车,并且还能知道它是不是新车。”张骊駻说。
未来,张骊駻团队期待这张全景图可以为天然药物的发现提供宏观地图和指南,从而促进对细菌中芳香聚酮的全面探索和开发。
图集欣赏:形态各异的放线菌,以及产生的芳香型聚酮化合物






本文来自微信公众号:西湖大学WestlakeUniversity(ID:westlakeuni),作者:张骊駻实验室,编辑:徐珊

