遗传变异图谱是研究人群演化史、医学遗传学、基因型-表型关联的基础。此前,大多数全基因组测序相关研究主要集中在欧洲血统人群。已有研究表明,罕见和低频的变异往往特定于人群或样本,尤其是与疾病相关的变异。中国人群全基因组测序资源和单倍型参考面板的缺乏阻碍了世界上最大人群的遗传学与精准医学研究。
为此,中国科学院院士、中科院生物物理研究所研究员徐涛团队,研究员何顺民团队合作,在Cell Reports上在线发表文章,介绍该团队关于"女娲"(NyuWa)中国人群基因组资源库的工作,提供针对中国人群的遗传变异图谱与参考面板基因型推演服务,旨在促进中国人群的遗传学与医学研究。
研究团队分析了2999个中国人的全基因组深度测序数据(26.2X),并以“女娲”命名。
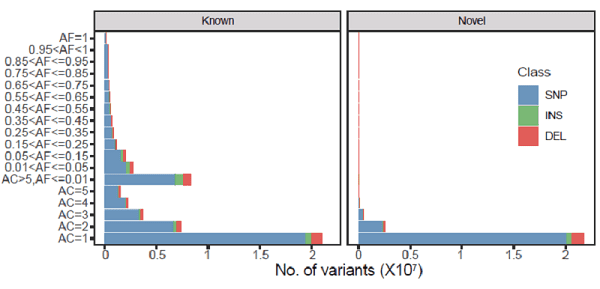
NyuWa全基因组测序资源的变异数量
基于NyuWa数据资源,研究构建了包含7106万SNPs和819万InDels的中国人群遗传变异图谱,并对其进行全面注释。相比其它人群队列,NyuWa数据集包含2501万新变异,其中包括14.9万非同义变异、10.1万有害变异、11493个编码和非编码基因的功能丧失变异、636个癌症相关基因的蛋白截短变异。
大量新变异表明,在以往遗传研究中,中国人群的变异代表性不足,NyuWa基因组资源则填补了这一空缺。
此外,根据临床相关数据库的注释,研究在NyuWa中发现了1140个致病变异,以及药物基因组学相关位点和癌症风险位点上中国人群与世界其他人群的变异频率差异。这些发现有助于中国人群精准医学研究,可能促进新的遗传学和医学进展。
与其他参考面板相比,NyuWa参考面板将汉族人群基因型推演的错误率降低了30%-51%,在大多数其他东亚和东北亚人群中也有优异表现。

