虚拟现实(VR)技术使科学家能够创建一个物体的3D模型,然后虚拟地“进去”查看,以更好地了解其结构和功能。这就是美国能源部(DOE)橡树岭国家实验室(ORNL)的科学家为研究引起COVID-19大流行的SARS-CoV-2病毒所做的事情。该团队使用中子和X射线绘制了新冠病毒的部分内部结构图,以创建一个精确的3D模型。
具体来说,科学家们绘制了主要的蛋白酶(Mpro),这是一种参与病毒复制的酶,他们在其中加入了一种使用高速计算机筛选发现的初步小分子。
利用VR技术观察酶模型,科学家们通过修改其结构虚拟构建了不同的小分子,以观察是否有任何新设计的化合物能够适合或结合到Mpro酶表面的一个关键部位。足够强的结合可以抑制或阻断该酶的功能,这对于阻止病毒在COVID-19患者体内繁殖至关重要。
为了确定特定的化学修饰对19种候选抑制剂与Mpro酶结合的紧密程度的影响,研究小组合成了每个抑制剂分子并测量了它们的结合强度。结合力越强,抑制剂就能更有效地阻止酶的运作和病毒的复制。
其中一个测试的抑制剂,即HL-3-68,与其他被测试的抑制剂相比,显示了与Mpro结合并抑制其功能的卓越能力。这项题为"抑制剂与SARS-CoV-2主要蛋白酶S1和S2子站点结合的结构、电子和静电决定因素"的研究细节发表在《药物化学杂志》上。
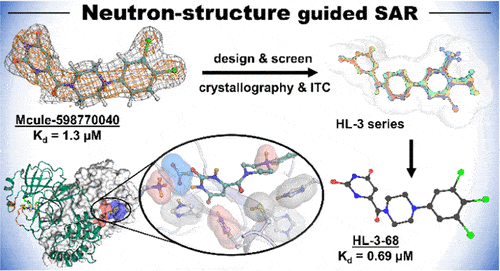
“我们的研究旨在更好地了解分子如何在Mpro酶的活性位点上结合,该酶在SARS-CoV-2复制中发挥着关键作用,”主要作者Daniel Kneller说。“在测试我们设计的分子的过程中,我们发现了一个含有一个额外的氯原子的分子,它显示出更大的抑制Mpro的能力。这种新的化学结构与全球社区以前研究的不同,可能会开辟新的研究途径,为防治SARS-CoV-2提供令人兴奋的可能性。”
Mpro酶上的活性位点与其他类型的新冠病毒相同,而且似乎不容易发生变异--这为可能设计一种对多种SARS-CoV-2变体和其他新冠病毒有效的抗病毒疗法提供了机会。
同样重要的是,该活性部位与人类酶中已知的活性部位不同,这将最大限度地减少可能导致患者产生副作用的非预期结合。
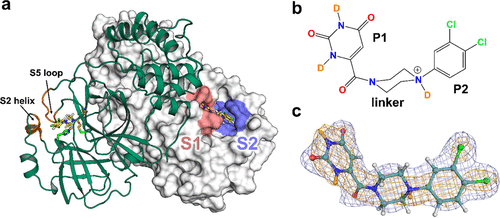
结构和分子生物学中心利用ORNL的散裂中子源(SNS)的设施和高通量同位素反应堆(HFIR)的资源,进行了Mpro酶样品的X射线测量和生产。候选抑制剂是由纳米相材料科学中心(CNMS)大分子纳米材料组的共同作者李辉和Peter Bonnesen合成的。
这项研究结合了大量的生物物理、生物化学和分子生物学方法,并包括虚拟现实辅助结构分析和小分子构建,汇集了来自ORNL、阿贡国家实验室、国家卫生研究院和田纳西大学诺克斯维尔分校的科学家。通讯作者Andrey Kovalevsky说:“这项研究的合作性质使我们能够发现小分子抑制剂在与该酶结合时必须遵守的规则,以便在药物设计和开发的漫长过程中对进一步的步骤有用。”
共同通讯作者Peter Bonnesen补充说:“对CNMS来说,这是一个新的和令人兴奋的项目,它利用了我们为用户合成定制有机分子的专业知识。在这个项目中,我们每次向我们的SNS同事提供几个候选分子。当有关分子作为抑制剂的有效性的结果出来后,团队将讨论对分子结构进行何种调整。然后,李辉和我将回到实验室,制造这些新的候选抑制剂。”

