本文来自微信公众号:果壳(ID:Guokr42),作者:栗子,编辑:麦麦,题图来自:unsplash
去年12月,有只名叫AlphaFold2的人工智能,在一个“困扰了生物学家50年”的问题上,获得了大突破——那就是蛋白结构预测问题。
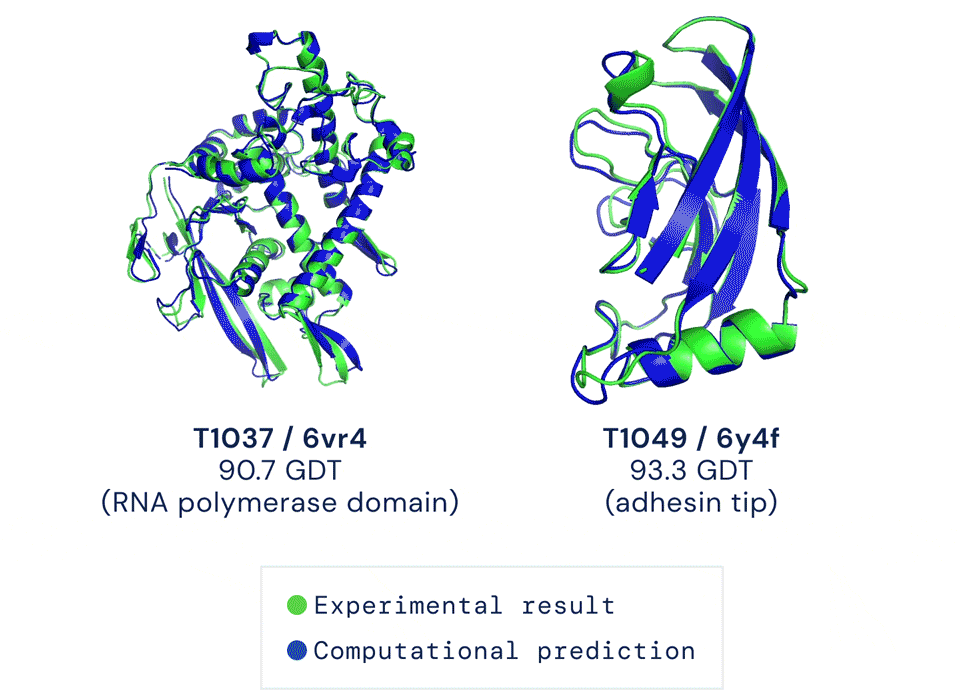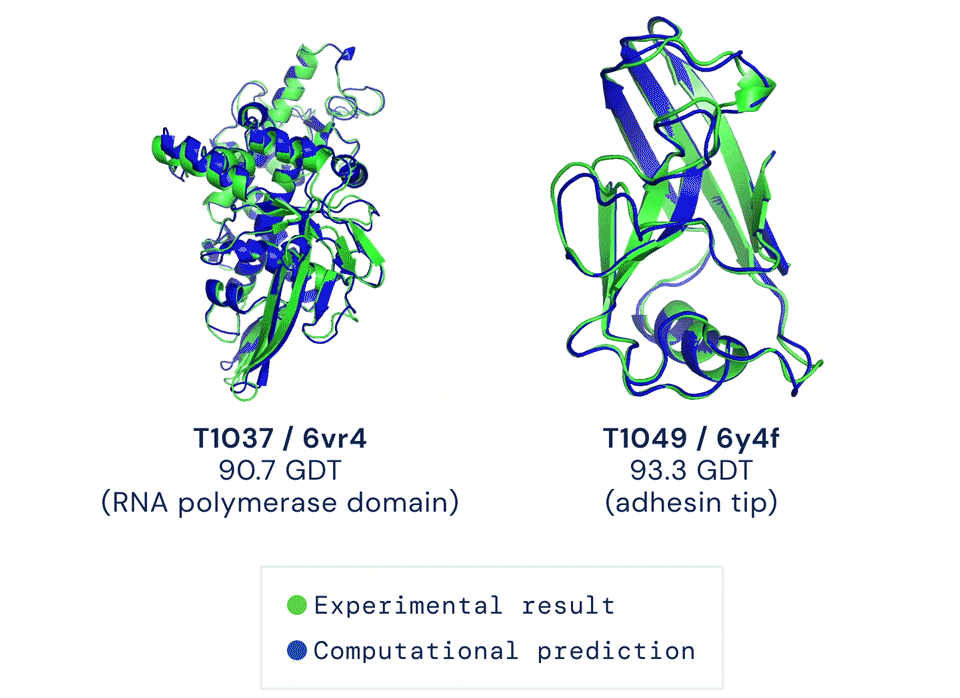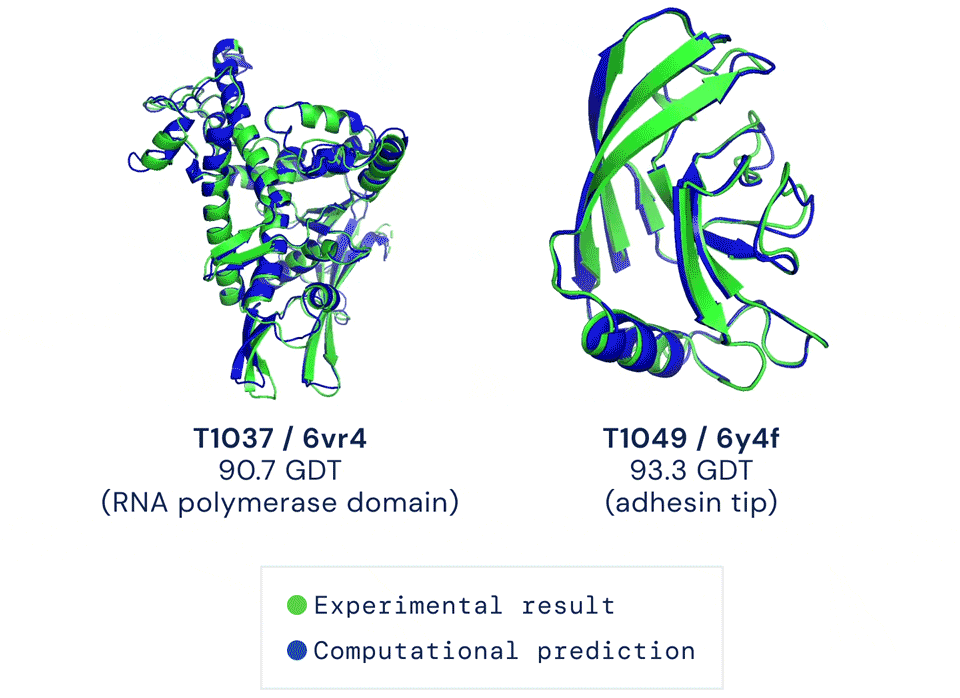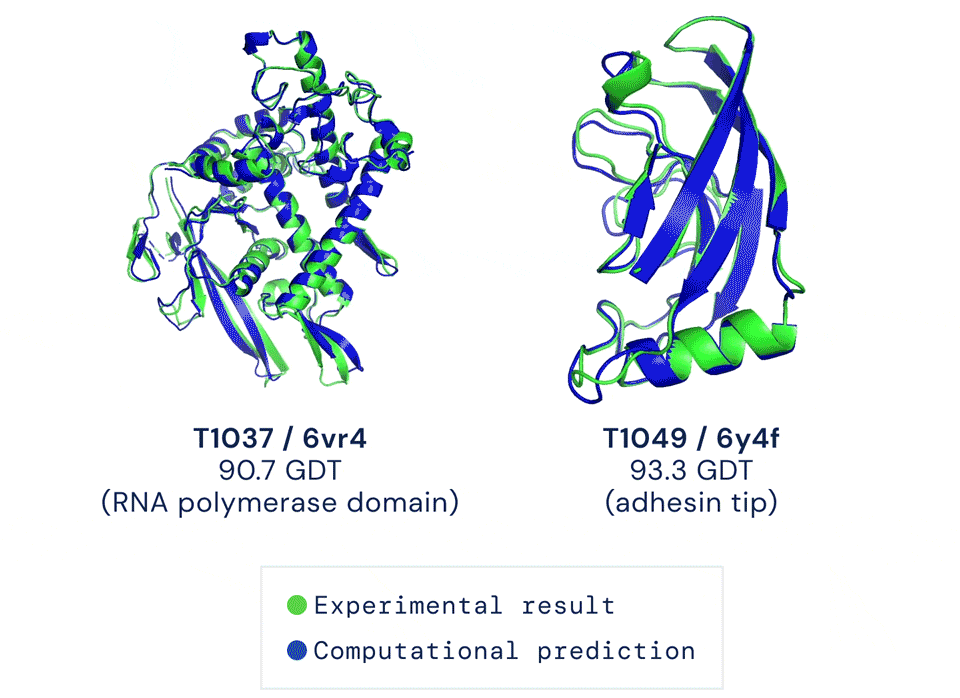
AlphaFold2以前所未有的预测准确度震撼了学界,Nature新闻标题也用“它会改变一切”来描述这只AI。只不过,一想到“这东西不会开源的”,人们不免有些沮丧。
但半年后,一只媲美AlphaFold2的开源AI诞生了。现在,AlphaFold2也开源了。
怎样预测结构
蛋白质是一串氨基酸折叠成的,但仅凭氨基酸的排列顺序、猜出它折叠后的三维结构,这一直都是难度很高的事情。
业界有一种十分有名的工具,名叫多序列对齐(MSA)。DeepMind团队开发AlphaFold2时也用到了它。
在物种演化的过程中,不同的动物如人类、鱼、兔子等等,身上有些蛋白质的功能是相似的,对应的氨基酸序列也会有不少相近之处。
假如把一些序列相近的蛋白质从数据集里搜出来,这些序列也许不一样长,也许有些位置发生了各自的变异,但只要把它们排到一起,在适当的位置插入空格,就可以让这些序列变得一样长,也可以让它们共有的部分对齐。

如此,便会有明显的规律跳出来:多个物种都保留下来的片段,可能就是体现蛋白质功能的区域。当然,保留不等于完全不变。
假设功能性片段里有个氨基酸变了,而蛋白质功能仍在,那应当还有另一个氨基酸跟着变了,这样两个氨基酸之间的相互作用才会保留,使功能不受影响。
所以,一旦发现两两一对的变化,AI很容易抓住特征,找到两个氨基酸之间的位置关系。
![2013年一项研究中,对齐后的成对氨基酸变化丨参考文献[3]<br label=图片备注 class=text-img-note>](https://i.aiapi.me/h/2021/07/19/Jul_19_2021_07_56_12_13679942451496377.png)
许多的类似的线索加在一起,最终能帮AlphaFold描绘出蛋白质的结构。
AI训练时,见过一些已知结构的蛋白质,和大量未知结构的氨基酸序列。而在训练完成后,就算已知结构的蛋白质中,没有一个和AI要预测的序列相近,它依然能完成预测,直接输出原子的三维坐标。
凭借这样的技能,AlphaFold2去年参加了蛋白结构预测挑战赛CASP14。结果,它的成绩远超所有对手,预测出的大部分结构与标准答案之间,大约只差一个原子的宽度。
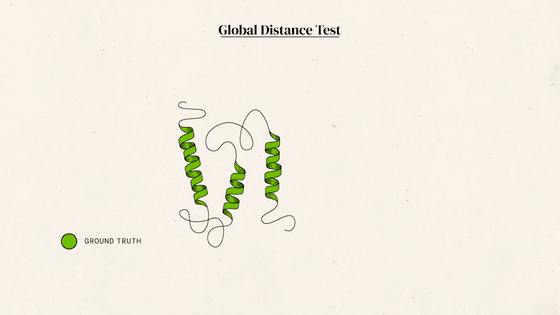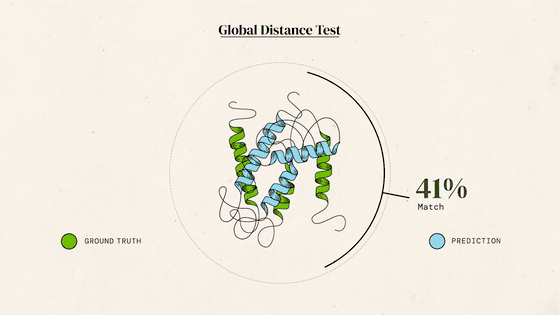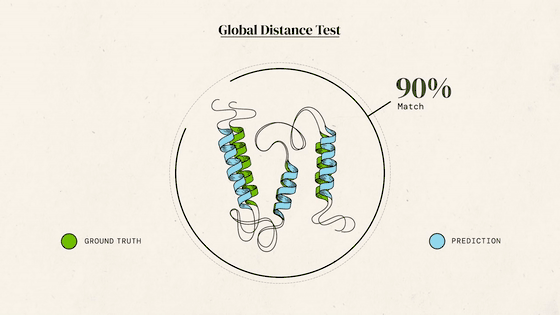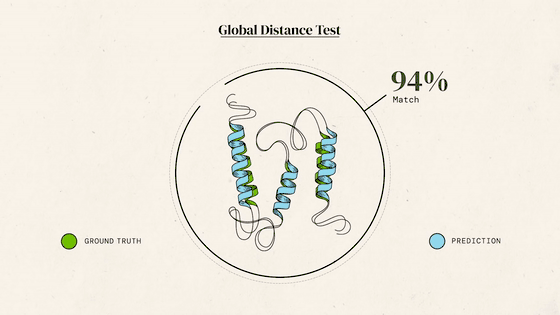
每道题满分100,通常得分超过90便认定答对。而AlphaFold2在所有题目里得分的中位数达到空前的92.4,答对了大约2/3的题目。而其他对手离90分还有不小的距离。
当然,也有些题目它答不对。开发团队在论文里提到了算法的一个不足,就是当数据集里找不到30个和目标序列相近的蛋白质(就是说连未知结构的相似序列都很少)来做对齐,预测效果会大打折扣。
你开源完我开源
赛后,DeepMind团队承诺将“分享我们的方法,并为科学共同体提供广泛、免费的获取途径”。而生物学家们盼了半年之后,昨天AlphaFold2论文终于在《自然》杂志上发表,代码也随之开源。
不过,这或许与隔壁家的一只AI有关。
同日,《科学》杂志也发表了一篇关于蛋白结构预测AI的论文。里面介绍的算法名叫RoseTTAFold,来自华盛顿大学,且已在6月15日开源。
而在这只AI开源后没几天,DeepMind首席执行官哈萨比斯便在推特更新了动态,说AlphaFold2论文正在接受评议,还说会开源代码,就快了。

说快也不假。AlphaFold2论文从被《自然》接收到上线,只经历了三天,发表前未经编辑,也不是在《自然》论文常规上线日期发出。对此,Science网站发布的蛋白结构预测AI新闻稿还提到:
Nature赶着发出这篇论文,就为了跟Science发的论文巧合一下。
如果说是刻意巧合,那么被追赶的RoseTTAFold又是一只怎样的AI?
它同样离不开多序列对齐(MSA)。不同的是它分三轨(three-track):一维的序列,二维的距离图(Distance Map),以及三维的坐标。信息在这三者间来回游走,AI推理的时候便能考虑到这三个层面之间的联系。
RoseTTAFold预测蛋白结构的准确度和AlphaFold2差不多,但并不像AlphaFold2那样需要好几台GPU的算力,一台RTX 2080就可以在10分钟左右完成一次蛋白质主链坐标的预测,长度大约400个氨基酸(这比去年比赛的AlphaFold2所需时间要短,不过开源版AlphaFold2速度已追上)。
![蛋白质复合体结构预测丨参考文献[2]<br label=图片备注 class=text-img-note>](https://i.aiapi.me/h/2021/07/19/Jul_19_2021_07_56_18_13679948913875950.png)
另外,AlphaFold2只预测过单个蛋白质的结构,而RoseTTAFold还尝试预测复合体——如果准确度足够高,这将会是更实用的技术,因为很多蛋白质的功能都离不开其与其他蛋白质的相互作用。
不过,RoseTTAFold的开发者之一大卫·贝克(David Baker)也坦言,他的工作灵感源于AlphaFold2,且自家AI预测准确度比AlphaFold2差一点点。
自开源至今,RoseTTAFold已经为用户预测了5000多个序列折叠后的蛋白结构。
而加州大学旧金山分校的结构生物学家大卫·阿加德(David Agard),在这只AI开源前便已成了测试版用户。他说,把序列发过去之后,几小时就拿到结果,节省了一年的时间。
阿加德和许多人一样,曾经在AlphaFold2横扫挑战赛的时候,感到兴奋又沮丧。
一方面,蛋白质的功能由三维结构决定,AI能准确地预测蛋白结构,意味着科学家们可以更好地预判某种蛋白质能不能和特定的分子结合,带来人们希望看到的反应。许多工作,如设计新药或新的酶,都可能因此而加速。
另一方面,DeepMind以不开源著称(大概),AlphaFold2官宣时的信息量只有一篇博客,人们甚至对它开源这件事并不抱希望,只是看着世界上存在这样的技术而自己用不到。
不过现在,生物学家们不仅能用上开源算法,还一下子有了两个。
参考文献
[1] Jumper, J., Evans, R., Pritzel, A., Green, T., Figurnov, M., Ronneberger, O., Tunyasuvunakool, K., Bates, R., ŽíDek, A., Potapenko, A., Bridgland, A., Meyer, C., Kohl, S. A. A., Ballard, A. J., Cowie, A., Romera-Paredes, B., Nikolov, S., Jain, R., Adler, J., . . . Hassabis, D. (2021). Highly accurate protein structure prediction with AlphaFold. Nature. Published. https://doi.org/10.1038/s41586-021-03819-2
[2] Baek, M., DiMaio, F., Anishchenko, I., Dauparas, J., Ovchinnikov, S., Lee, G. R., Wang, J., Cong, Q., Kinch, L. N., Schaeffer, R. D., Millán, C., Park, H., Adams, C., Glassman, C. R., DeGiovanni, A., Pereira, J. H., Rodrigues, A. V., van Dijk, A. A., Ebrecht, A. C., . . . Baker, D. (2021). Accurate prediction of protein structures and interactions using a three-track neural network. Science. Published. https://doi.org/10.1126/science.abj8754
[3] Ekeberg, M., Lövkvist, C., Lan, Y., Weigt, M., & Aurell, E. (2013). Improved contact prediction in proteins: using pseudolikelihoods to infer Potts models. Physical Review E, 87(1), 012707.
[4] Researchers unveil ‘phenomenal’ new AI for predicting protein. (2021, July 15). Science | AAAS. https://www.sciencemag.org/news/2021/07/researchers-unveil-phenomenal-new-ai-predicting-protein-structures
[5] Ewen Callaway (2021, July 15). DeepMind’s AI for protein structure is coming to the masses. Nature. https://www.nature.com/articles/d41586-021-01968-y
[6] The AlphaFold Team. (2020, November 30). AlphaFold: a solution to a 50-year-old grand challenge in biology. DeepMind. https://deepmind.com/blog/article/alphafold-a-solution-to-a-50-year-old-grand-challenge-in-biology
[7] Senior, A. W., Evans, R., Jumper, J., Kirkpatrick, J., Sifre, L., Green, T., ... & Hassabis, D. (2020). Improved protein structure prediction using potentials from deep learning. Nature, 577(7792), 706-710.
本文来自微信公众号:果壳(ID:Guokr42),作者:栗子

